Transformador de columna¶
Introducción¶
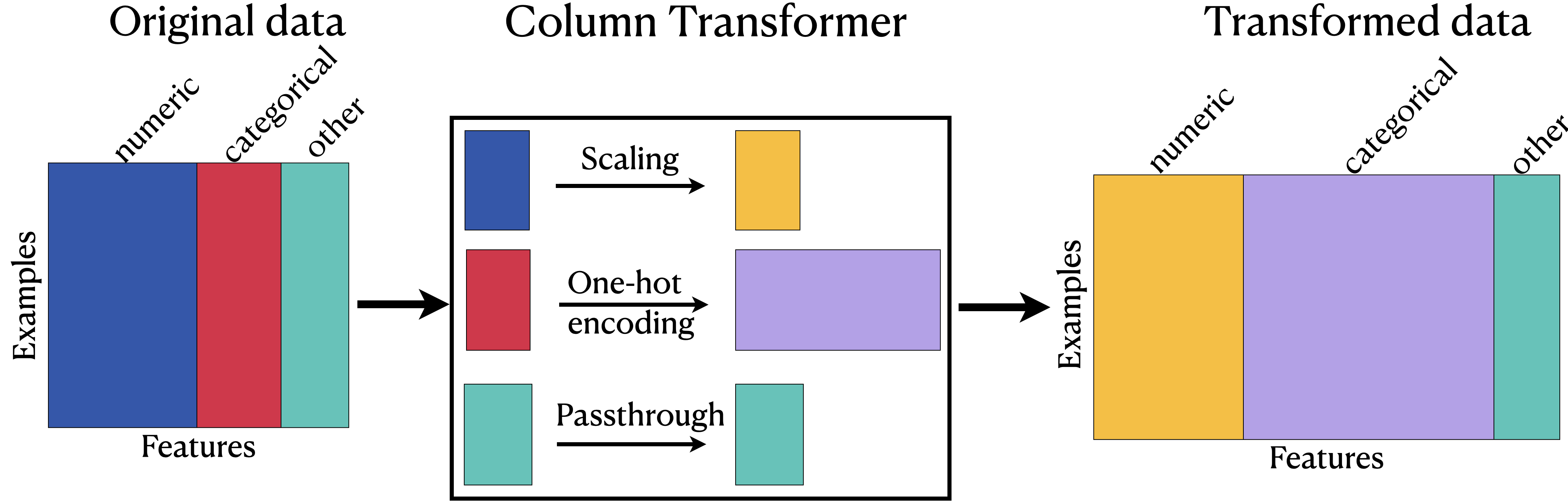
Pueden ver que el transformador de columna funciona en paralelo. Escala los datos numéricos, realiza una codificación one-hot en los datos categóricos y todo lo demás que no está en esas categorías se pasa al conjunto de datos inalterado final. Estos pasos ocurren al mismo tiempo.
También observen que las columnas categóricas son ahora más largas de lo que eran antes del transformador de columna. Esto se debe porque cuando se realiza una codificación one-hot, hay una columna separada que se creó para cada categoría en cada columna categórica.
Al usar un transformador de columna les permitirá aplicar diferentes tipos de transformador a diferentes columnas en los datos. Es conveniente y permite ver fácilmente qué transformaciones se aplicaron a qué columnas durante la fase de preprocesamiento.
Ejemplo
Este ejemplo utiliza el conjunto de datos médicos. Este conjunto de datos muestra una serie de información sobre el paciente, en particular, su información socioeconómica junto con la condición médica que les diagnosticaron.
La “Codificación ordinal y OneHotEncoder”, se mostró como seleccionar las características del objeto y ponelas en un DataFrame separado para la codificación one-hot. Luego tuvieron que unir el DataFrame de codificación one-hot con las columnas numéricas. En este ejemplo, verán cómo realizar diferentes transformaciones de procesamiento a diferentes características en los datos en un paso usando un transformador de columna. Simultáneamente:
- Escalarán columnas numéricas usando Standard Scaler.
- Realizarán una codificación one-hot a las columnas categóricas usando OneHotEncoder.
# importaciones
import pandas as pd
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.compose import make_column_selector
from sklearn.preprocessing import StandardScaler, OneHotEncoder
# leer datos
df = pd.read_csv('data/medical_data.csv') # or your path here
df.head()
| State | Lat | Lng | Area | Children | Age | Income | Marital | Gender | ReAdmis | ... | Hyperlipidemia | BackPain | Anxiety | Allergic_rhinitis | Reflux_esophagitis | Asthma | Services | Initial_days | TotalCharge | Additional_charges | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | AL | 34.34960 | -86.72508 | Suburban | 1.0 | 53 | 86575.93 | Divorced | Male | 0 | ... | 0.0 | 1.0 | 1.0 | 1.0 | 0 | 1 | Blood Work | 10.585770 | 3726.702860 | 17939.403420 |
| 1 | FL | 30.84513 | -85.22907 | Urban | 3.0 | 51 | 46805.99 | Married | Female | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 1 | 0 | Intravenous | 15.129562 | 4193.190458 | 17612.998120 |
| 2 | SD | 43.54321 | -96.63772 | Suburban | 3.0 | 53 | 14370.14 | Widowed | Female | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 0 | 0 | Blood Work | 4.772177 | 2434.234222 | 17505.192460 |
| 3 | MN | 43.89744 | -93.51479 | Suburban | 0.0 | 78 | 39741.49 | Married | Male | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 1 | 1 | Blood Work | 1.714879 | 2127.830423 | 12993.437350 |
| 4 | VA | 37.59894 | -76.88958 | Rural | 1.0 | 22 | 1209.56 | Widowed | Female | 0 | ... | 1.0 | 0.0 | 0.0 | 1.0 | 0 | 0 | CT Scan | 1.254807 | 2113.073274 | 3716.525786 |
5 rows × 32 columns
# Formato para ML y train test split
X = df.drop(columns = 'Additional_charges')
y = df['Additional_charges']
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 42)
Realizar selectores de columnas
Estamos haciendo dos selectores: uno para los datos del objeto y otro para los datos de los números.
# Instanciar los selectores de columnas categóricas y numéricas para seleccionar las columnas adecuadas
cat_selector = make_column_selector(dtype_include='object')
num_selector = make_column_selector(dtype_include='number')
Instanciar los transformadores
Ahora que han realizado los selectores, necesitan instanciar cada uno de los transformadores de columnas que quieran usar. Ambos son procesadores comunes que a menudo usarán en el procesamiento. Un StandardScaler para escalar las columnas numéricas y un OneHotEncoder para codificar las columnas categóricas.
Nota: Matrices dispersas vs. arrays densos
Fíjense que en el siguiente código que no estén usando el argumento sparse=False en el constructor de OneHotEncoder. Las clases de modelo que usarán después pueden procesar bien las matrices dispersas, y estas suelen ser necesarias para conservar memoria cuando el DataFrame resultante es muy largo. En ejemplos anteriores establecimos sparse=False debido a que los arrays vacíos son difíciles de interpretar visualmente y no se pueden transformar fácilmente en un DataFrames de Pandas.
El ColumnTransformer utilizado posteriormente devuelve la matriz dispersa a un array denso internamente cuando la concatena con los datos numéricos escalados durante la transformación.
# Instanciar el escalador estándar y el codificador one hot
scaler = StandardScaler()
ohe = OneHotEncoder(sparse_output=False, handle_unknown='ignore')
Emparejar el transformador con las columnas
El siguiente paso se trata de emparejar las columnas con la transformación que queremos aplicar. Vamos a escalar los valores numéricos para nuestros números usando Standard Scaler. Vamos a confidicar one-hot cualquier valor categórico para nuestras columnas categóricas usando la codificación one-hot. Los vamos a emparejar al crear tuplas donde el primer elemento es el transformador y el segundo elemento es una lista de columnas o un objeto ColumnSelector.
# Make tuples for preprocessing the categorical and numeric columns
num_tuple = (scaler, num_selector)
cat_tuple = (ohe, cat_selector)
Instanciación de ColumnTransformer
Ahora tenemos de instanciar el transformador de columna y agregar cada una de las tuplas que creamos antes. Observen que tenemos un nuevo import para obtener make_column_transformer.
from sklearn.compose import make_column_transformer
col_transformer = make_column_transformer(num_tuple, cat_tuple, remainder = 'passthrough')
# Encajar el transformador
col_transformer.fit(X_train)
ColumnTransformer(remainder='passthrough',
transformers=[('standardscaler', StandardScaler(),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A30>),
('onehotencoder',
OneHotEncoder(handle_unknown='ignore',
sparse_output=False),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A60>)])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
ColumnTransformer(remainder='passthrough',
transformers=[('standardscaler', StandardScaler(),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A30>),
('onehotencoder',
OneHotEncoder(handle_unknown='ignore',
sparse_output=False),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A60>)])<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A30>
StandardScaler()
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F1684E5A60>
OneHotEncoder(handle_unknown='ignore', sparse_output=False)
[]
passthrough
# Transformación
X_train_processed = col_transformer.transform(X_train)
X_test_processed = col_transformer.transform(X_test)
# Ver las transformaciones
X_train_df = pd.DataFrame(X_train_processed)
X_train_df.head()
| 0 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | ... | 97 | 98 | 99 | 100 | 101 | 102 | 103 | 104 | 105 | 106 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | -0.508205 | 0.281935 | -0.065104 | 0.272586 | -1.123467 | 0.0 | 0.509399 | -0.008943 | 0.014639 | -0.620174 | ... | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 1 | -0.720642 | 0.252836 | 1.235813 | 1.119125 | -0.619881 | 0.0 | -0.999823 | 1.907372 | 0.014639 | -0.620174 | ... | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2 | -0.493403 | 0.482823 | -0.498743 | 0.272586 | -0.518276 | 0.0 | 0.119354 | -0.967100 | 0.014639 | -0.620174 | ... | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 3 | 1.134821 | -0.434666 | 2.103090 | 1.307245 | 1.938720 | 0.0 | -1.044875 | -0.008943 | -0.983474 | 2.762592 | ... | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 4 | 0.587322 | 0.497439 | -0.932382 | -0.809103 | -0.328240 | 0.0 | 1.283708 | -0.008943 | 0.014639 | -0.620174 | ... | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
5 rows × 107 columns
X_train_df.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 750 entries, 0 to 749 Columns: 107 entries, 0 to 106 dtypes: float64(107) memory usage: 627.1 KB
Ahora todas nuestras transformaciones están completas y están listas para ser modeladas. Podemos ver que las columnas numéricas se han escalado y las columnas categóricas se han codificado como números. Todas las columnas aparecen ahora como columnas numéricas y están listas para el aprendizaje automático. ¡Misión cumplida!
SimpleImputer¶
SimpleImputer es una clase en la biblioteca scikit-learn de Python que se utiliza para completar valores faltantes o nulos en un conjunto de datos. Cuando trabajas con conjuntos de datos reales, es común encontrar valores faltantes en algunas columnas, lo que puede dificultar el análisis y el modelado de datos.
SimpleImputer proporciona una forma sencilla de manejar estos valores faltantes al reemplazarlos por valores predeterminados o estadísticamente calculados. Puedes especificar cómo deseas completar los valores faltantes utilizando diferentes estrategias.
Algunas de las estrategias de imputación que se pueden utilizar con SimpleImputer son:
mean(media): reemplaza los valores faltantes con la media de la columna en la que se encuentran.median(mediana): reemplaza los valores faltantes con la mediana de la columna.most_frequent(más frecuente): reemplaza los valores faltantes con el valor más frecuente en la columna.constant(constante): reemplaza los valores faltantes con un valor constante especificado por el usuario.
Estas estrategias permiten imputar los valores faltantes de forma rápida y sencilla antes de realizar análisis o entrenar modelos de aprendizaje automático en scikit-learn.
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.compose import make_column_selector, make_column_transformer
from sklearn.impute import SimpleImputer
from sklearn import set_config
set_config(display='diagram')
# leer datos
df = pd.read_csv("data/medical_data.csv")
df.head()
| State | Lat | Lng | Area | Children | Age | Income | Marital | Gender | ReAdmis | ... | Hyperlipidemia | BackPain | Anxiety | Allergic_rhinitis | Reflux_esophagitis | Asthma | Services | Initial_days | TotalCharge | Additional_charges | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | AL | 34.34960 | -86.72508 | Suburban | 1.0 | 53 | 86575.93 | Divorced | Male | 0 | ... | 0.0 | 1.0 | 1.0 | 1.0 | 0 | 1 | Blood Work | 10.585770 | 3726.702860 | 17939.403420 |
| 1 | FL | 30.84513 | -85.22907 | Urban | 3.0 | 51 | 46805.99 | Married | Female | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 1 | 0 | Intravenous | 15.129562 | 4193.190458 | 17612.998120 |
| 2 | SD | 43.54321 | -96.63772 | Suburban | 3.0 | 53 | 14370.14 | Widowed | Female | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 0 | 0 | Blood Work | 4.772177 | 2434.234222 | 17505.192460 |
| 3 | MN | 43.89744 | -93.51479 | Suburban | 0.0 | 78 | 39741.49 | Married | Male | 0 | ... | 0.0 | 0.0 | 0.0 | 0.0 | 1 | 1 | Blood Work | 1.714879 | 2127.830423 | 12993.437350 |
| 4 | VA | 37.59894 | -76.88958 | Rural | 1.0 | 22 | 1209.56 | Widowed | Female | 0 | ... | 1.0 | 0.0 | 0.0 | 1.0 | 0 | 0 | CT Scan | 1.254807 | 2113.073274 | 3716.525786 |
5 rows × 32 columns
# Examinar los valores faltantes
df.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 1000 entries, 0 to 999 Data columns (total 32 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 State 995 non-null object 1 Lat 1000 non-null float64 2 Lng 1000 non-null float64 3 Area 995 non-null object 4 Children 993 non-null float64 5 Age 1000 non-null int64 6 Income 1000 non-null float64 7 Marital 995 non-null object 8 Gender 995 non-null object 9 ReAdmis 1000 non-null int64 10 VitD_levels 1000 non-null float64 11 Doc_visits 1000 non-null int64 12 Full_meals_eaten 1000 non-null int64 13 vitD_supp 1000 non-null int64 14 Soft_drink 1000 non-null int64 15 Initial_admin 995 non-null object 16 HighBlood 1000 non-null int64 17 Stroke 1000 non-null int64 18 Complication_risk 995 non-null object 19 Overweight 1000 non-null int64 20 Arthritis 994 non-null float64 21 Diabetes 994 non-null float64 22 Hyperlipidemia 998 non-null float64 23 BackPain 992 non-null float64 24 Anxiety 998 non-null float64 25 Allergic_rhinitis 994 non-null float64 26 Reflux_esophagitis 1000 non-null int64 27 Asthma 1000 non-null int64 28 Services 995 non-null object 29 Initial_days 1000 non-null float64 30 TotalCharge 1000 non-null float64 31 Additional_charges 1000 non-null float64 dtypes: float64(14), int64(11), object(7) memory usage: 250.1+ KB
print(df.isna().sum().sum(), 'missing values')
72 missing values
Hay en total 72 valores que faltan repartidos en 14 columnas diferentes.
Algunas columnas con datos faltantes son numéricas y otras son categóricas (objeto). Podemos usar mean, median, mode, most_frequent o estrategias de imputación (imputer) constante para los datos numéricos, pero solo las estrategias constante o most_frequent para los datos categóricos
Nota: Si este fuera un proyecto real, podríamos investigar más para ver si deberíamos eliminar filas o columnas con datos faltantes, o imputar los datos faltantes.
df[df.isna().any(axis=1)].shape
> (70, 32)
Faltan 70 filas de 100 en al menos un valor. Eso es el 0,7 % de nuestros datos. En un proyecto real podríamos solo eliminar las filas con valores faltantes con df.dropna().
La imputación de los valores faltantes puede filtrar información de los datos de prueba a los datos de entrenamiento, así que imputamos los valores DESPUÉS de dividir los datos
# train/test split
X = df.drop(columns=['Additional_charges'])
y = df['Additional_charges']
# Train Test Split
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
Vamos a separar nuestras características en dos tipos de columnas basándonos en el tipo de datos. Una será nuestras columnas que incluirán los números enteros y flotantes. La otra columna será las columnas categóricas que incluyen nuestras cadenas (objetos).
# instancien los selectores a nuestros tipos de datos numéricos y categóricos
num_selector = make_column_selector(dtype_include='number')
cat_selector = make_column_selector(dtype_include='object')
# seleccionen las columnas numéricas de cada tipo
num_columns = num_selector(X_train)
cat_columns = cat_selector(X_train)
# comprueben las listas
print('numeric columns are:\n', num_columns)
numeric columns are: ['Lat', 'Lng', 'Children', 'Age', 'Income', 'ReAdmis', 'VitD_levels', 'Doc_visits', 'Full_meals_eaten', 'vitD_supp', 'Soft_drink', 'HighBlood', 'Stroke', 'Overweight', 'Arthritis', 'Diabetes', 'Hyperlipidemia', 'BackPain', 'Anxiety', 'Allergic_rhinitis', 'Reflux_esophagitis', 'Asthma', 'Initial_days', 'TotalCharge']
# comprueben las listas
print('categorical columns are:\n', cat_columns)
categorical columns are: ['State', 'Area', 'Marital', 'Gender', 'Initial_admin', 'Complication_risk', 'Services']
Antes que decidamos cuál estrategia utilizar para la imputación, necesitamos comprender nuestros datos. El código de abajo aislarán las columnas numéricas a las que les faltan datos. Podemos hacer esto para ver qué estrategia de imputación deberíamos usar.
# aíslen las columnas numéricas
df_num = df[num_columns]
# aíslen las columnas con datos faltantes
df_num.loc[:, df_num.isna().any()]
| Children | Arthritis | Diabetes | Hyperlipidemia | BackPain | Anxiety | Allergic_rhinitis | |
|---|---|---|---|---|---|---|---|
| 0 | 1.0 | 1.0 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 |
| 1 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2 | 3.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 3 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 4 | 1.0 | 0.0 | NaN | 1.0 | 0.0 | 0.0 | 1.0 |
| ... | ... | ... | ... | ... | ... | ... | ... |
| 995 | 3.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 996 | 2.0 | 1.0 | 0.0 | NaN | 1.0 | 1.0 | 1.0 |
| 997 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 |
| 998 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 999 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 |
1000 rows × 7 columns
Todas las columnas numéricas parecen tener valores enteros (aunque no son tipos de datos enteros). Si utilizáramos una estrategia de "mean", se rellenarían con valores decimales (flotantes). Para rellenarlos con valores enteros necesitamos usar estrategias de “median”.
En primer lugar, vamos a comprobar a qué columnas le faltan datos.
X_train.isna().any()
State True Lat False Lng False Area True Children True Age False Income False Marital True Gender True ReAdmis False VitD_levels False Doc_visits False Full_meals_eaten False vitD_supp False Soft_drink False Initial_admin True HighBlood False Stroke False Complication_risk True Overweight False Arthritis True Diabetes True Hyperlipidemia True BackPain True Anxiety True Allergic_rhinitis True Reflux_esophagitis False Asthma False Services True Initial_days False TotalCharge False dtype: bool
El siguiente código muestra cómo aplicar el Simple Imputer a las columnas que fueron previamente seleccionadas como y definidas como num_columns. ¡Tengan en cuenta que el paso con .fit solo se aplica al conjunto de entrenamiento!
# Instancien el objeto imputer de la clase SimpleImputer con la estrategia 'median'
median_imputer = SimpleImputer(strategy='median')
# Encajen el objeto imputer en los datos de entrenamiento numérico con .fit()
# calculen las medianas (medians) de las columnas en el conjunto de entrenamiento
median_imputer.fit(X_train[num_columns])
#Utilicen la mediana a partir de los datos de entrenamiento para rellenar los valores que en falten
#las columnas numericas de los conjuntos de entrenamiento y de prueba con .transform()
X_train.loc[:, num_columns] = median_imputer.transform(X_train[num_columns])
X_test.loc[:, num_columns] = median_imputer.transform(X_test[num_columns])
¿Rellenó SimpleImputer los valores faltantes en X_train?
X_train.isna().any()
State True Lat False Lng False Area True Children False Age False Income False Marital True Gender True ReAdmis False VitD_levels False Doc_visits False Full_meals_eaten False vitD_supp False Soft_drink False Initial_admin True HighBlood False Stroke False Complication_risk True Overweight False Arthritis False Diabetes False Hyperlipidemia False BackPain False Anxiety False Allergic_rhinitis False Reflux_esophagitis False Asthma False Services True Initial_days False TotalCharge False dtype: bool
Las columnas numéricas no tienen valores faltantes, pero las categóricas las siguen teniendo.
Recreemos nuestro X_train original con todos los fatos faltantes y veamos cómo ColumnTransformer, combinado con SimpleImputer, puede imputar las columnas numéricas con medianas, y las columnas categóricas y el valor más frecuente.
# Train Test Split
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=42)
X_train.isna().any()
State True Lat False Lng False Area True Children True Age False Income False Marital True Gender True ReAdmis False VitD_levels False Doc_visits False Full_meals_eaten False vitD_supp False Soft_drink False Initial_admin True HighBlood False Stroke False Complication_risk True Overweight False Arthritis True Diabetes True Hyperlipidemia True BackPain True Anxiety True Allergic_rhinitis True Reflux_esophagitis False Asthma False Services True Initial_days False TotalCharge False dtype: bool
Ambas columnas categóricas y numéricas tienen valores faltantes.
# instancien los selectores a nuestros tipos de datos numéricos y categóricos
num_selector = make_column_selector(dtype_include='number')
cat_selector = make_column_selector(dtype_include='object')
Rellenaremos los datos faltantes en columnas numéricas con “median” de cada columna y datos faltantes en columnas categóricas con el valor más frecuente. Estas no son las únicas opciones, pero es lo que haremos hoy.
# Instanciar SimpleImputers con estrategias most_frequent y median
freq_imputer = SimpleImputer(strategy='most_frequent')
median_imputer = SimpleImputer(strategy='median')
Como podrán recordar de las otras clases, make_column_transformer() toma las tuplas de la forma (transformador, columnas). ColumnSelectors se puede usar en vez de las listas de columnas. Los dos son aceptables. Podemos establecer remainder='passthrough' si no aplicamos los transformadores a todas las columnas. Podríamos hacerlo si ya hubiéramos imputado algunas columnas a mano.
El valor por defecto para ColumnTransformer es eliminar cualquier columna que no especificada en una tupla, mientras que remainder = 'passthrough' retiene los valores originales para alguna columna no especificada en una tupla sin ninguna transformación.
# creen tuplas de (imputer, selector) para cada tipo de dato
num_tuple = (median_imputer, num_selector)
cat_tuple = (freq_imputer, cat_selector)
# instanciación de ColumnTransformer
col_transformer = make_column_transformer(num_tuple, cat_tuple, remainder='passthrough')
col_transformer
ColumnTransformer(remainder='passthrough',
transformers=[('simpleimputer-1',
SimpleImputer(strategy='median'),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EAB3D0>),
('simpleimputer-2',
SimpleImputer(strategy='most_frequent'),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EABEB0>)])In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. On GitHub, the HTML representation is unable to render, please try loading this page with nbviewer.org.
ColumnTransformer(remainder='passthrough',
transformers=[('simpleimputer-1',
SimpleImputer(strategy='median'),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EAB3D0>),
('simpleimputer-2',
SimpleImputer(strategy='most_frequent'),
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EABEB0>)])<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EAB3D0>
SimpleImputer(strategy='median')
<sklearn.compose._column_transformer.make_column_selector object at 0x000001F168EABEB0>
SimpleImputer(strategy='most_frequent')
passthrough
# ajustar ColumnTransformer en los datos de entrenamiento
col_transformer.fit(X_train)
# transformen los datos de entrenamiento y de prueba (esto generará un array de NumPy)
X_train_imputed = col_transformer.transform(X_train)
X_test_imputed = col_transformer.transform(X_test)
# cambien el resultado regreso a un DataFrame
X_train_imputed = pd.DataFrame(X_train_imputed, columns=X_train.columns)
X_train_imputed.isna().any()
State False Lat False Lng False Area False Children False Age False Income False Marital False Gender False ReAdmis False VitD_levels False Doc_visits False Full_meals_eaten False vitD_supp False Soft_drink False Initial_admin False HighBlood False Stroke False Complication_risk False Overweight False Arthritis False Diabetes False Hyperlipidemia False BackPain False Anxiety False Allergic_rhinitis False Reflux_esophagitis False Asthma False Services False Initial_days False TotalCharge False dtype: bool